2022年10月17日,我院蒋庆华课题组在英国牛津大学出版社《Nucleic Acids Research》期刊发表最新研究成果《DeepST: identifying spatial domains in spatial transcriptomics by deep learning》,提出了一种高精度的空间域(在基因表达和组织形态学上具有相似性的区域)识别算法DeepST,它是一种基于深度学习的网络集成算法,通过在大量不同分辨率的空间转录组学数据集上进行测试,显示DeepST的空间域识别性能优于已经公开发表的空间域识别方法。

1 简介
复杂组织的功能与不同细胞类型的空间分布有关。组织中转录表达的相对位置对于了解其生物学功能至关重要。空间转录组学(ST)的突破性技术,如10× Visium、Slideseq和Stereoseq,已经能够在捕获位置(称为斑点)以几个细胞甚至亚细胞水平的分辨率对基因表达进行全基因组分析。
识别空间域(即在基因表达和组织学上具有相似性的一个区域)是空间转录组学研究中面临的巨大挑战之一。目前,识别空间域的方法主要分为两类:非空间聚类方法和空间聚类方法。传统的非空间聚类算法,如K-means和Louvain,将基因表达数据作为输入,通常它们的聚类与组织切片几乎不对应。空间聚类方法主要结合基因表达、空间位置和形态学,以解释基因表达的空间依赖性,从而更好地匹配空间位置,如BayesSpace、stLearn、SpaGCN等。虽然这些算法可以将斑点或细胞识别为不同的区域,但它们主要依靠线性主成分分析来提取基因表达的高度可变特征,主要涉及线性变换,因此它们无法建模复杂的非线性相互作用。此外,大多数方法不能整合不同批次的ST数据,并且它们无法处理其他空间组学数据,这使得它们的通用性降低。
为了解决这些挑战,作者研发了基于深度学习的空间域识别技术(DeepST)。DeepST使用预先训练好的深度神经网络模型从组织形态学信息(H&E染色图片)中提取图像特征,然后将提取的特征与基因表达和空间位置数据集成,以表征空间相邻点的相关性,并创建空间增强的基因表达矩阵。DeepST使用图神经网络(GNN)编码器和去噪自编码器联合生成增强ST数据的潜在表示,域对抗神经网络(DAN)用于整合来自多个批次或不同技术平台的ST数据。
为了证明DeepST在空间域识别方面的性能,作者对不同平台(10×Visium、SlideseqV2和Stereoseq)生成的ST数据作为基准,与现有算法进行了广泛测试和比较。DeepST被应用到其他空间组学数据上,如4i和MIBI-TOF,特别是在MERFISH数据上提取三维(3D)表达域。在乳腺癌组织中,识别出了传统方法无法检测出的视觉同质肿瘤区域内的异质亚区域,得到了更精细的空间结构域划分。实验结果表明,该技术在空间域识别方面展现出强大的能力,同样在处理其他空间组学数据方面也具有很强的扩展性。
2 结果
2.1 DeepST工作流程概述
DeepST通过整合基因表达、空间位置和组织形态学信息的低维嵌入来表征空间域(图1A)。首先通过预先训练的深度学习网络处理来自H&E染色的形态学图片,建立形态学特征矩阵。结合形态特征和空间邻近信息,实现对斑点的基因表达增强(图1B)。然后,使用去噪自编码器学习从集成特征空间到低维表示空间的线性映射,以减少模型过拟合。同时,DeepST通过k近邻方法计算空间坐标的图邻接矩阵。将一个变分图自动编码器插入同一模型框架中,以绘制点的空间关联,从而通过与相应空间相邻点的集成表示生成空间嵌入(图1C)。 除了空间域识别任务,作者还在DeepST中实现了整合多个空间平台数据或去批次的算法,它主要是将潜在嵌入递送入由梯度反转层连接的域鉴别器中(图1C)。总而言之,该算法得到的潜在嵌入可用于识别空间域、纠正批次效应和执行各种下游分析。
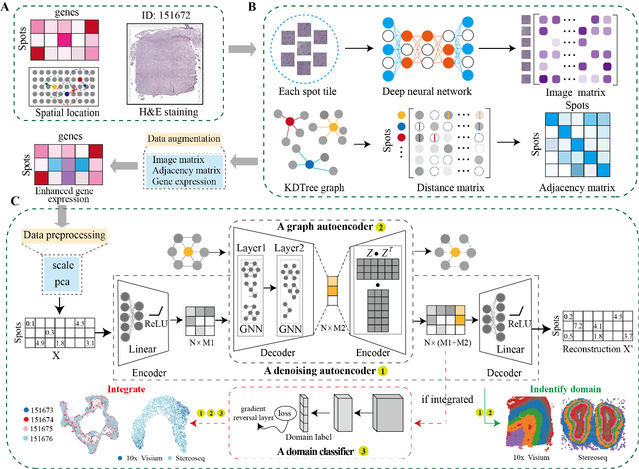
图 1 DeepST算法的工作流程
2.2 基准测试
为了测试DeepST在识别空间域算法方面的性能,作者在12张人脑背外侧前额叶皮质(DLPFC,图2A)上进行了基准测试。主要比较了包括非空间和空间算法在内的7种算法。结果表明DeepST识别的空间域与DLPFC的手动注释和神经科学中皮质分层的定义是一致的(图2B, 2C)。DeepST算法的调整兰德指数(ARI)为0.515+/0.011,高于当前最佳算法(BayesSpace,ARI=0.463+/0.012;Wilcoxon检验,P值=0.007)。DeepST在编号151671切片上获得了最佳聚类精度(ARI=0.798)。在151673切片上,DeepST和BayesSpace成功描绘了L1和L2皮质层,这是任何其他方法都无法识别到的(图2B)。
作者进一步评估了DeepST在更为精细划分的小鼠脑组织上的空间域识别能力,DeepST识别出小鼠大脑中海马区的角状回和齿状回部分(图2F),以及后矢状面中的小脑皮质和背状回(图2F)区域,这与参考注释一致。作者同样也注意到,空间域的个数设置往往影响模型的性能。因此,作者设计了自适应计算最佳聚类分辨率的方法,并在小鼠大脑后部切片中获得较好的聚类得分。作者同样在算法性能上进行了比较,DeepST处理大约4,000个点和30,000个基因的空间转录组数据,需要大约7分钟(在GPU上运行)和大约6G内存,而BayesSpace需要大约四倍于DeepST的时间和更高的内存使用率。
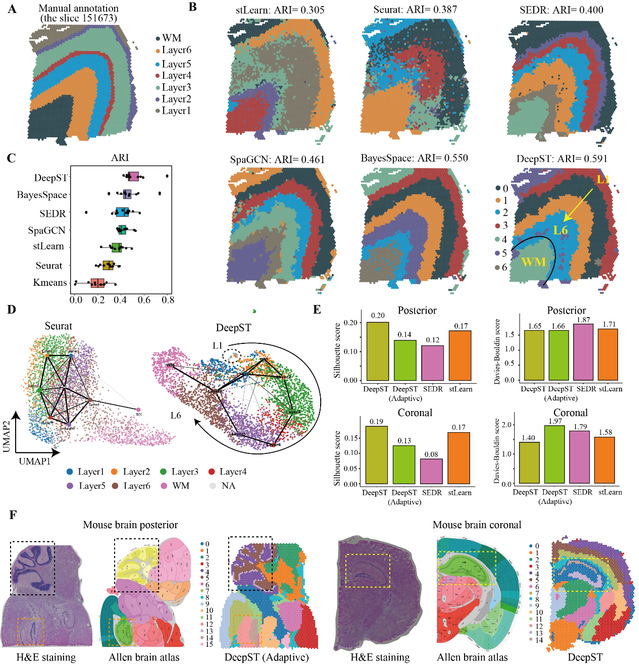
图 2 DeepST提高脑组织的空间域识别能力
2.3 DeepST更精细地从癌症组织中识别空间域
为了更好的验证DeepST在癌症组织上的泛化能力,作者继续测试了一组乳腺癌的空间转录组数据。整体上,DeepST识别的空间域与手动注释的结构域高度一致(图 3A、3B)。与其他的空间算法识别的空间域相比,DeepST的空间域具有更高的区域连续性和更少的噪声点(图3C)。作者设置了10和20个空间域,发现肿瘤区域,随着参数域变大而划分地更为精细,但健康区域仍保持原有的域结构。当K=20时,肿瘤内部区域出现了不同的异质区域,如域4和13(图3B)。作者对这两个空间域进行了差异表达分析,发现显著的差异基因(DEGs)ABCC11、ABCC12和TFF1与耐药和肿瘤分化相关(图3E)。作者同样对域1和域4进行了差异表达分析和通路富集分析,共检测到298个DEGs。作者发现域1可能是癌生长受促炎症免疫反应限制的区域(图3F),而域4表现出更强的肿瘤进展和转移,以及与化疗耐药相关(图3H)。总而言之,DeepST在刻画精细空间结构域方面表现出了卓越的性能。
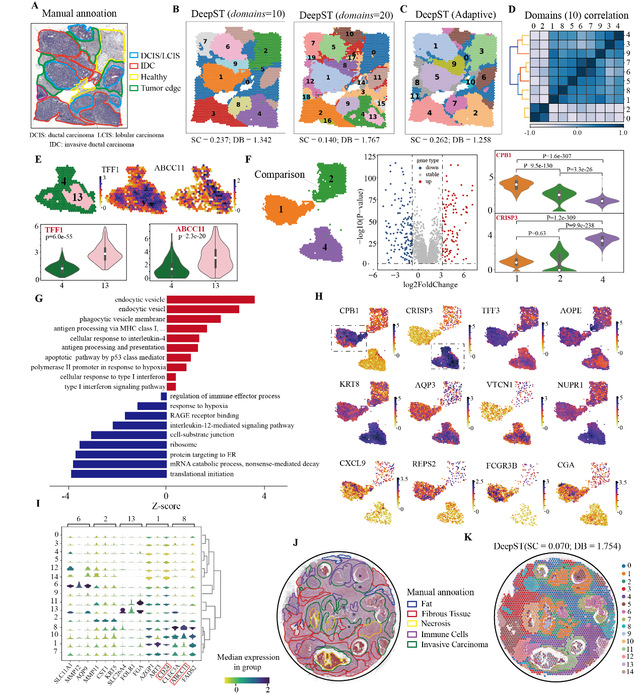
图3 DeepST可以更精细地从癌症组织中识别空间域
2.4 DeepST应用于其他空间组学数据
除了10×Genomics Visium平台之外,作者测试了DeepST在基于成像的分子数据(MERFISH、4i和MIBI-TOF)和高分辨率ST数据(Stereoseq和SlideseqV2)的泛化能力。作者首先将DeepST应用于4i(迭代间接免疫荧光成像)数据,该数据测量了从毫米到纳米尺度(约270,000个观察值/像素)的高通量生物样本中的40个蛋白质读数。DeepST比SEDR和stLearn更详细地描述了局部区域的亚细胞分布,包括每个细胞内的各种隔室、细胞器和细胞结构(图4A)。在另一个基于成像的分子MIBI-TOF数据上,DeepST在四个成像结果上显示了部分区域连续性和局部元素融合性(图4C)。
作者在接近单细胞分辨率的小鼠海马SlideseqV2数据(41,786个亚细胞和4,000个基因)上测试DeepST识别空间域的性能。相比较于其他的空间算法,DeepST的空间域显示出更强的区域连续性,如“DentatePyramids”和“Endolifeal_Tip”结构域(图 4B)。作者强调了DeepST可用于识别3D结构域,而大多数空间算法可能无法处理这些3D数据。DeepST整合了三个连续批次的小鼠视前下丘脑MERFISH数据,清晰地解析了“Ependymal”和“OD Mature”3D结构域,批次处理提供了比单个空间域识别更清晰的3D分子结构分布(图4D)。作者还在小鼠小鼠嗅球Stereoseq芯片数据集上,测试了识别空间域和整合不同空转数据(10×Genomics Visium和Stereoseq)的能力。DeepST准确地识别了嗅神经层、内丛状层、肾小球层、二尖瓣细胞层、颗粒细胞层和外丛状层,且与已知的解剖特征相匹配(图4F)。最后DeepST整合后的空间域显示出比SEDR、Harmony和Scanorama更大的域融合(图4G),且保留了更大的生物学变异。

图4 DeeST应用于各种空间组学数据
3 总结
在这项研究中,作者开发了一个深度学习框架,它整合了空间位置、组织形态学和基因表达信息,识别具有基因表达和组织形态学相似的空间域。DeepST不仅可以准确识别空间域和纠正批次效应,还可以适用于其他不同的ST技术平台,如MERFISH、Slide-seq和Stereoseq。同样,DeepST也显示了处理其他空间组学数据的潜力(4i和MIBI-TOF)。应用于乳腺癌ST数据集,DeepST可以更精细地划分肿瘤组织中的亚区域。随着更多ST数据的生成,DeepST有望成为破译空间细胞分布新原理的强有力工具。
蒋庆华教授为该论文的通讯作者,博士研究生徐昌、靳喜云、韦淞仁为并列第一作者,该研究得到了国家自然科学基金委、哈尔滨工业大学青年科学家工作室的支持。
论文链接
https://academic.oup.com/nar/article-lookup/doi/10.1093/nar/gkac901
论文资料
Xu C, Jin X, Wei S, Wang P, Luo M, Xu Z, Yang W, Cai Y, Xiao L, Lin X, Liu H, Cheng R, Pang F, Chen R, Su X, Hu Y, Wang G, Jiang Q*. DeepST: identifying spatial domains in spatial transcriptomics by deep learning. Nucleic Acids Res. 2022 Oct 17;gkac901. doi: 10.1093/nar/gkac901.
数据链接:
http://spatial.libd.org/spatialLIBD
https://support.10xgenomics.com/spatial-gene-expression/datasets
https://github.com/scverse/squidpy
https://github.com/BGIResearch/stereopy
https://www.spatialomics.org/SpatialDB/
代码链接:
https://github.com/JiangBioLab/DeepST

